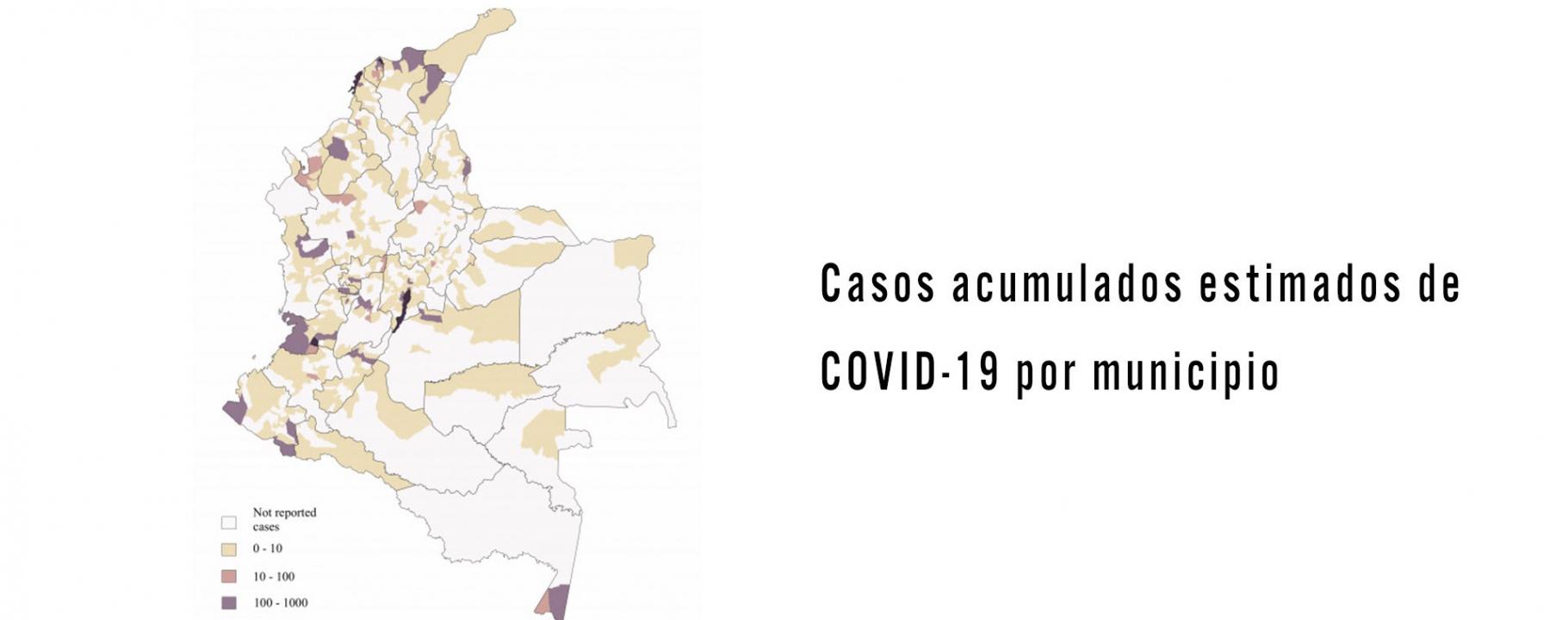
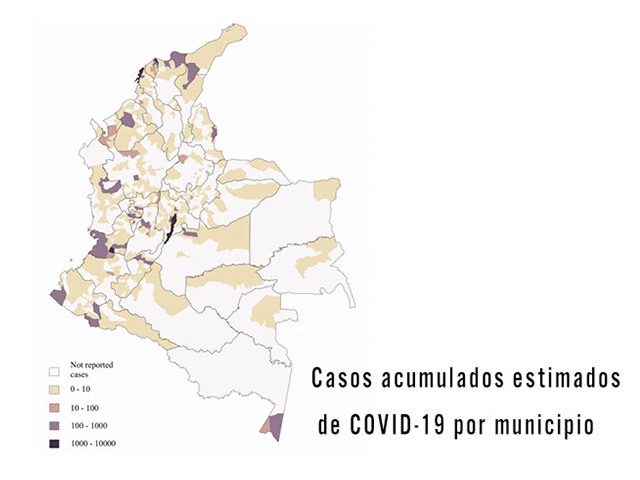
Desde el 6 de marzo de 2020, fecha en que se reportó el primer caso del nuevo Coronavirus en Colombia, el virus se ha propagado rápidamente a varios municipios del país (hasta el 09 julio se reportaron casos en 555 municipios). El 20 de marzo, el Gobierno decretó cuarentena obligatoria a nivel nacional para prevenir la propagación del virus. Sin embargo, en la actualidad las medidas de la cuarentena se han relajado y el país está comenzando a reactivarse. Por lo tanto, determinar en tiempo real la variación en la transmisión de la enfermedad en las diferentes regiones del país es esencial para evaluar los posibles efectos de su reapertura. Realizar pronósticos eficaces sirve para la detección temprana de los lugares y los periodos en el tiempo que tendrán alta intensidad de transmisión; información vital para que el sistema de salud pública intervenga de forma efectiva.
Integrantes del grupo de investigación de Biología Matemática y Computacional (BIOMAC) del Departamento de Ingeniería Biomédica de la Universidad de los Andes, desarrollaron un modelo matemático que, basado en datos de movilidad humana, pronostica la dinámica de la transmisión del SARS-COV2 entre diferentes ciudades y municipios del país. Este modelo es capaz de rastrear la evolución del brote, estimar la dinámica de los parámetros epidemiológicos en el tiempo y capturar la variación geográfica de la incidencia de la enfermedad. Además, puede ser utilizado para predecir el comportamiento de la enfermedad en diferentes escenarios; por lo cual, es una herramienta valiosa para desarrollar un sistema de vigilancia en tiempo real para la gestión y control del COVID-19.
El modelo desarrollado por los investigadores del BIOMAC puede proyectar y pronosticar los casos diarios de la enfermedad en los diferentes municipios del país. Este modelo identifica poblaciones susceptibles, expuestas, asintomáticas, infectadas y recuperadas de cada municipio y periodo en el tiempo. Además, utiliza tres supuestos epidemiológicos principales sobre la propagación del SARS-CoV2: i) Asume el retraso de la infección a la fecha del informe, esta suposición evalúa directamente el desafío de reconstruir la serie temporal de nuevas infecciones, ya que las observaciones ocurren mucho después del momento de transmisión. ii) Estima las tasas de contacto variables en el tiempo, tanto para individuos diagnosticados como para infecciones asintomáticas o leves, que explican directamente las restricciones de movilidad impuestas para reducir la transmisión. iii) Supone una fracción de infecciones asintomáticas o leves variables en el tiempo, que explica el posible alto número de infecciones asintomáticas en el país.
Los investigadores alimentaron el modelo con los datos de infecciones diarias reportados por el Instituto Nacional de Salud Colombiano (INS) y con los datos de movimiento regulares de Facebook (el número de los viajes que los usuarios de Facebook realizan entre ubicaciones a lo largo del tiempo). Los datos epidemiológicos usados también incluyen la fecha de recuperación (cuando el resultado es negativo para el virus), fecha de fallecimiento, edad, sexo, municipio, departamento, tipo (importado de otro país versus adquirido localmente), ubicación (si el paciente se encuentra actualmente en su hogar, hospital o en una Unidad de Cuidados Intensivos) y el estado de la enfermedad (síntomas leves, medios o severos). El modelo ha revelado que la tasa de infecciones no registradas en el país es del 45%.
Los integrantes del Departamento de Ingeniería Biomédica autores de este modelo son: Juan Manuel Cordovez, director y profesor asociado; Mauricio Santos-Vega, investigador postdoctoral; Carlos Bravo, estudiante doctoral y Jaime Cascante, estudiante de Maestría.
Noticias relacionadas:
El día despúes: la post-cuarentena
COVID-19: efecto de las medidas de mitigación en los próximos meses